上一篇简单的介绍了COX生存分析结果绘制森林图Forest plot(森林图) | Cox生存分析可视化,本文将介绍根据数据集合的基本信息以及点估计值(置信区间区间)的结果直接绘制森林图的方法。
其中点估计值(置信区间)的结果可以是COX也可以是logistic回归等其他方法的结果,适用范围更广。
数据准备
#载入R包
library(forestplot)
#数据来源:https://www.r-bloggers.com/forest-plot-with-horizontal-bands/
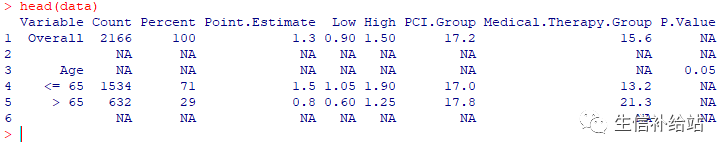
data <- read.csv("ForestPlotData.csv", stringsAsFactors=FALSE)
#查看数据
head(data)
绘制森林图
2.1 简单森林图
对数据进行部分修改,方便行名和列名字输出
## 构建tabletext,更改列名称,展示更多信息
np <- ifelse(!is.na(data$Count), paste(data$Count," (",data$Percent,")",sep=""), NA)
## The rest of the columns in the table.
tabletext <- cbind(c("Subgroup","n",data$Variable),
c("No. of Patients (%)","n",np),
c("4-Yr Cum. Event Raten PCI","n",data$PCI.Group),
c("4-Yr Cum. Event Raten Medical Therapy","n",data$Medical.Therapy.Group),
c("P Value","n",data$P.Value))
##绘制森林图
forestplot(labeltext=tabletext, graph.pos=3,
mean=c(NA,NA,data$Point.Estimate),
lower=c(NA,NA,data$Low), upper=c(NA,NA,data$High),
boxsize=0.5)
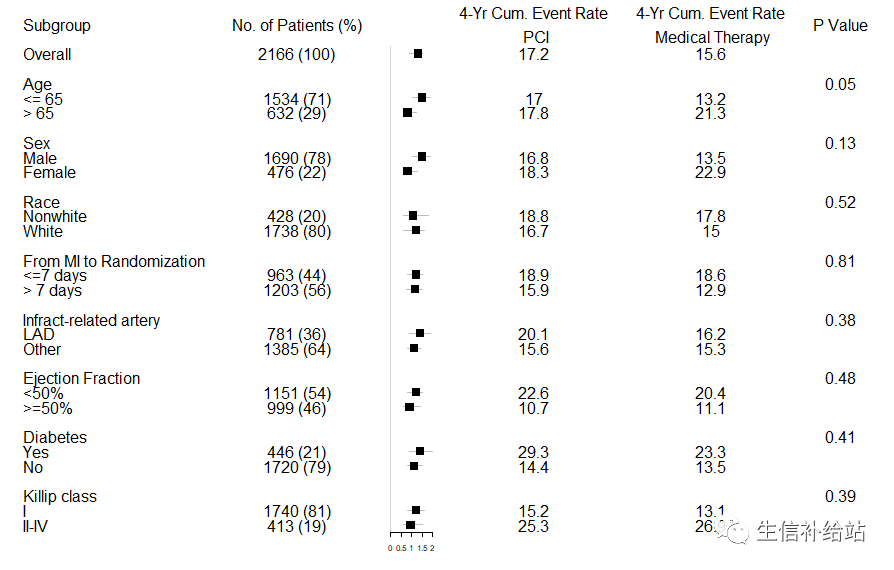
如上图所示基本信息OK了,但是可以在以下几个方面进行优化:
-
添加线条,区分Subgroup
-
更改箱线图的宽度,颜色和大小
-
更改字体大小,更易区分
-
添加标题和横坐标轴标示
2.2 优化森林图
## 定义亚组,方便后面线条区分
subgps <- c(4,5,8,9,12,13,16,17,20,21,24,25,28,29,32,33)
data$Variable[subgps] <- paste(" ",data$Variable[subgps])
forestplot(labeltext=tabletext,
graph.pos=3, #为Pvalue箱线图所在的位置
mean=c(NA,NA,data$Point.Estimate),
lower=c(NA,NA,data$Low), upper=c(NA,NA,data$High),
#定义标题
title="Hazard Ratio Plot",
##定义x轴
xlab=" <---PCI Better--- ---Medical Therapy Better--->",
##根据亚组的位置,设置线型,宽度造成“区块感”
hrzl_lines=list("3" = gpar(lwd=1, col="#99999922"),
"7" = gpar(lwd=60, lineend="butt", columns=c(2:6), col="#99999922"),
"15" = gpar(lwd=60, lineend="butt", columns=c(2:6), col="#99999922"),
"23" = gpar(lwd=60, lineend="butt", columns=c(2:6), col="#99999922"),
"31" = gpar(lwd=60, lineend="butt", columns=c(2:6), col="#99999922")),
#fpTxtGp函数中的cex参数设置各个组件的大小
txt_gp=fpTxtGp(label=gpar(cex=1.25),
ticks=gpar(cex=1.1),
xlab=gpar(cex = 1.2),
title=gpar(cex = 1.2)),
##fpColors函数设置颜色
col=fpColors(box="#1c61b6", lines="#1c61b6", zero = "gray50"),
#箱线图中基准线的位置
zero=1,
cex=0.9, lineheight = "auto",
colgap=unit(8,"mm"),
#箱子大小,线的宽度
lwd.ci=2, boxsize=0.5,
#箱线图两端添加小竖线,高度
ci.vertices=TRUE, ci.vertices.height = 0.4)
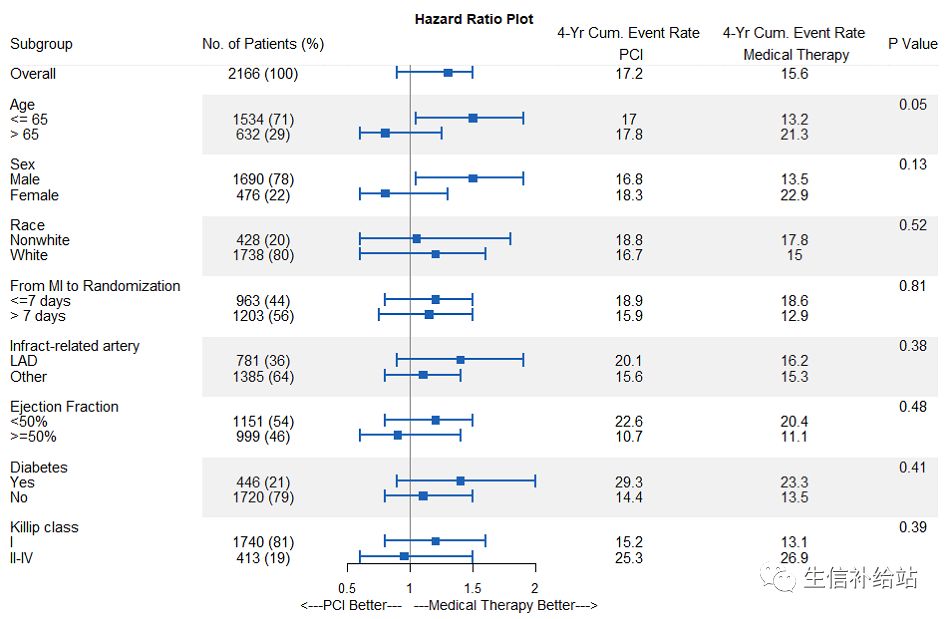
如此即绘制完成了,颜色,大小,间隔等需要根据实际情况进行调整。

更多:
R|生存分析(1):生存分析介绍以及绘制KM曲线
Nomogram(诺莫图) | Logistic、Cox生存分析结果可视化
maftools | 从头开始绘制发表级oncoplot(瀑布图)
ggalluvial|炫酷桑基图(Sankey),你也可以秀

长按上方二维码获即可关注本号~
本篇文章来源于微信公众号: OncoLab
 微信扫一扫打赏
微信扫一扫打赏
 支付宝扫一扫打赏
支付宝扫一扫打赏