杂志名称:Cell
发表日期:2024.01.25
DOI:10.1016/j.cell.2024.01.008.
PubMed:https://pubmed.ncbi.nlm.nih.gov/38301646/
影响因子:64.5
内容简介
基因组不稳定性可以触发促进肿瘤排斥的癌症内源性天然免疫应答。然而,癌细胞经常通过过表达免疫检查点调节剂(如PD-L1)来逃避这些应答。在这里,我们发现SNF2家族DNA转位酶SMARCAL1是一个促进肿瘤免疫逃逸的关键因子,它通过抑制天然免疫信号和诱导PD-L1介导的免疫检查点应答的双重机制发挥作用。在机制上,SMARCAL1限制了内源性DNA损伤,从而在癌细胞生长过程中抑制了cGAS-STING依赖的信号传导。同时,它与AP-1家族成员JUN合作,维持PD-L1转录调控元件上的染色质可访问性,从而促进癌细胞中PD-L1的表达。SMARCAL1的丧失阻碍了肿瘤细胞在基因组不稳定性反应中诱导PD-L1的能力,增强了抗肿瘤免疫应答,并在小鼠黑色素瘤模型中使肿瘤对免疫检查点阻断敏感。总的来说,这些研究揭示了SMARCAL1是癌症免疫治疗的一个有潜力的靶点。
创新性及研究意义
这篇文章的核心创新性在于发现了SMARCAL1这一蛋白质在促进肿瘤免疫逃逸中的双重调节作用。研究指出SMARCAL1通过两种机制促进肿瘤的免疫逃逸:一是抑制癌细胞内天然免疫信号的激活,二是促进PD-L1(一种免疫检查点蛋白)的表达,从而抑制免疫系统对肿瘤的攻击。此外,研究还发现,SMARCAL1的缺失能够增强抗肿瘤免疫应答,并在小鼠黑色素瘤模型中提高对免疫检查点阻断治疗的敏感性。
研究意义方面,这项工作揭示了SMARCAL1作为肿瘤免疫调节的关键因子,为癌症免疫治疗提供了新的靶点。这一发现不仅深化了我们对肿瘤免疫逃逸机制的理解,也为开发针对SMARCAL1的新型癌症治疗策略提供了理论基础。
数据图
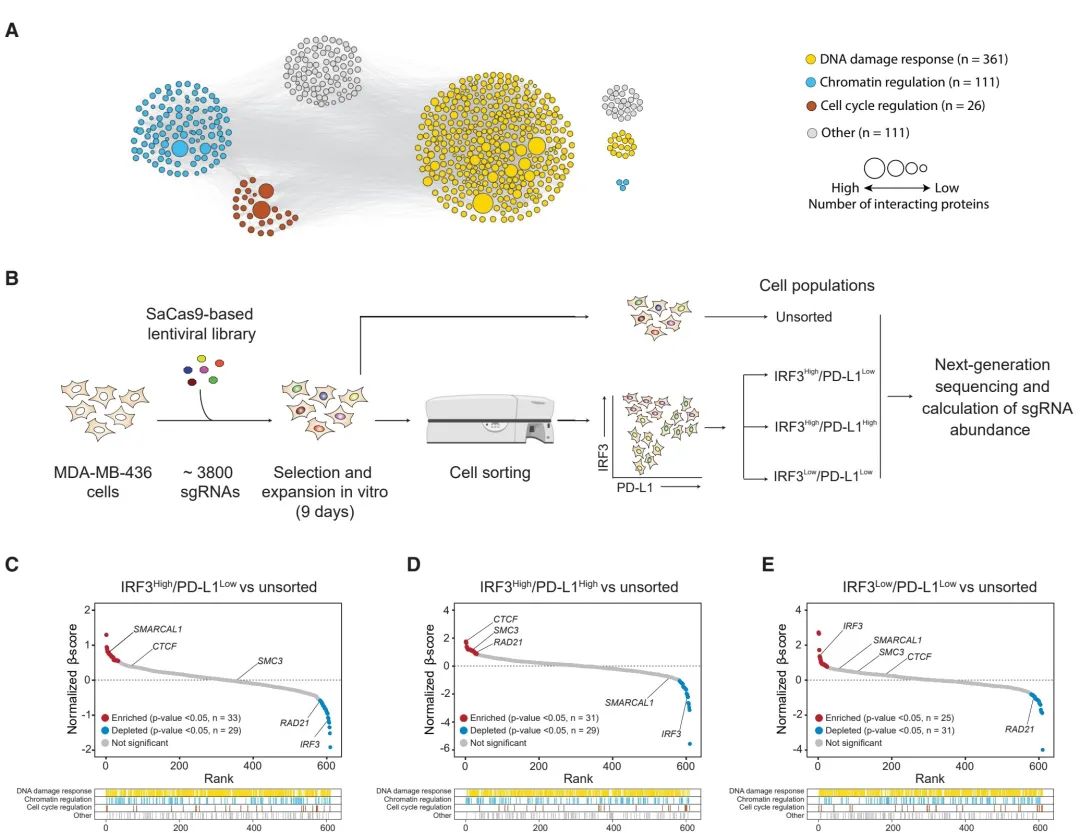
图1:在MDA-MB-436三阴性乳腺癌细胞中应用FACS的CRISPR-Cas9基因筛选研究
(A) sgRNA文库1针对的基因网络。节点(基因)的大小表示根据PrePPI数据,各节点(基因)之间的遗传和物理交互作用(灰色连线)的频次。
(B) 识别核内IRF3和PD-L1调节因子的CRISPR筛选流程图。在选取经慢病毒sgRNA文库1转导的MDA-MB-436细胞后,将细胞整体收集(未分选)或根据指标分为三个亚群。通过高通量测序技术确定不同细胞亚群中sgRNA的相对丰度。
(C–E) 根据检测组中各sgRNA的相对丰度,对sgRNA文库1目标基因进行排名,并展示其标准化β分数的分布。使用未分选的细胞群作为参照,通过MAGeCK最大似然估计法(MLE)计算得到IRF3高表达/PD-L1低表达(C)、IRF3高表达/PD-L1高表达(D)和IRF3低表达/PD-L1低表达(E)亚群中基因的标准化β分数。红色和蓝色点分别代表与sgRNA丰度增加和减少相关(β分数正值或负值,p < 0.05)的基因。
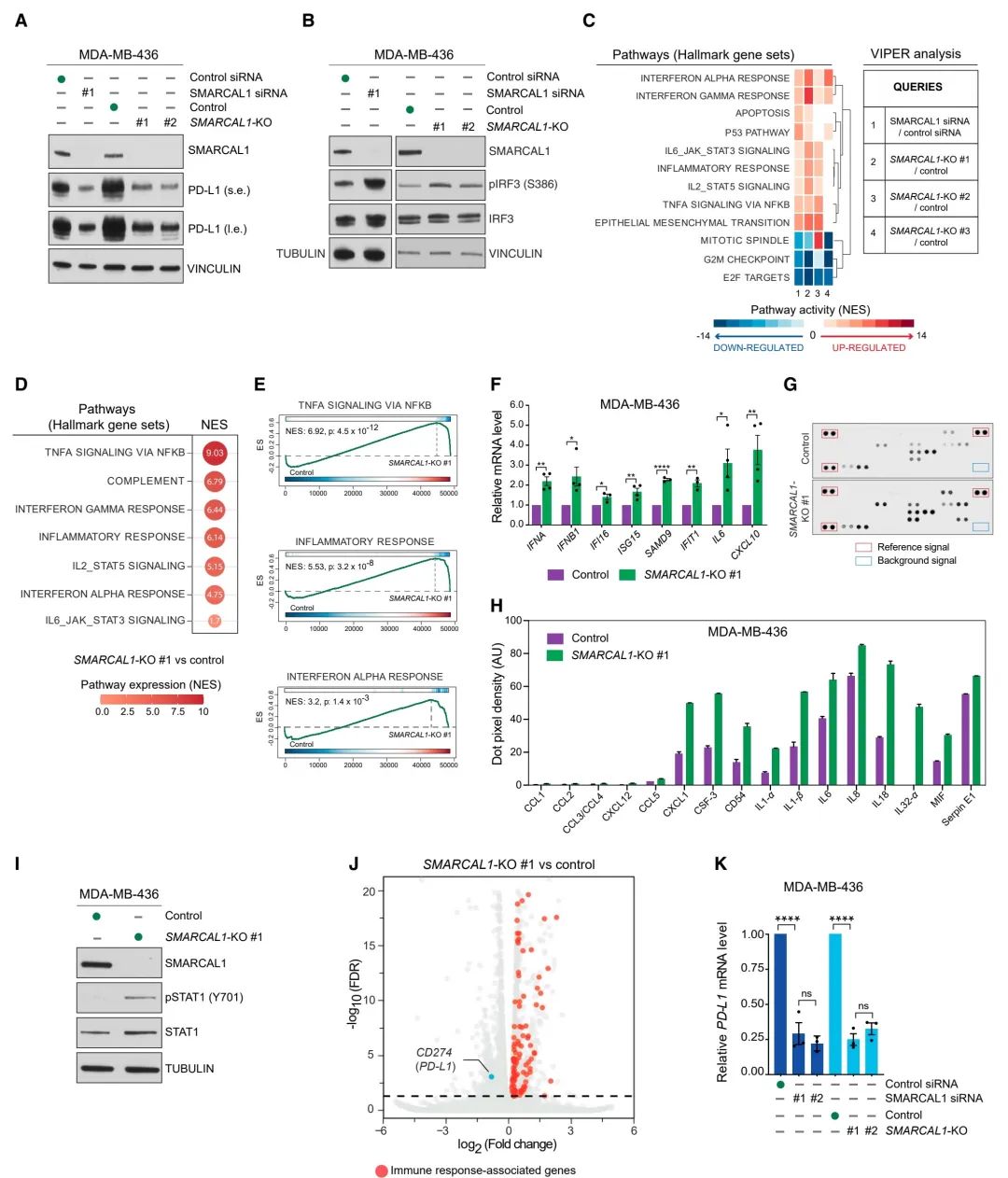
图2:SMARCAL1缺陷下的PD-L1表达及癌细胞固有先天免疫分析
(A) 免疫印迹显示了用指定siRNA处理的MDA-MB-436细胞、MDA-MB-436对照细胞或SMARCAL1- KO克隆中SMARCAL1、PD-L1和vinculin的水平。s.e.表示短曝光;l.e.表示长曝光。
(B) 免疫印迹图显示了在(A)中描述的细胞系中SMARCAL1、磷酸化IRF3(pIRF3 S386)、整体IRF3,以及tubulin或vinculin的水平。
(C) 相对于对照MDA-MB-436细胞,通过VIPER确定SMARCAL1缺失细胞(Query 1)和SMARCAL1-KO克隆(Query 2-4)在指定的标志基因集(MSigDB)内的活性评分。
(D) 与对照MDA-MB-436细胞相比,SMARCAL1-KO克隆#1在标志基因集(MSigDB)中的标准化富集评分(NESs)。
(E) 对照MDA-MB-436细胞,SMARCAL1-KO克隆#1在指定的标志基因集(MSigDB)中的基因集富集分析。
(F) 实时定量PCR(RT-qPCR)分析对照组及SMARCAL1-KO #1 MDA-MB-436细胞中IFNA、IFNB1、选定的干扰素刺激基因(ISGs)和促炎细胞因子的mRNA水平。数据展示了在SMARCAL1-KO #1与对照细胞中基因表达的倍数变化。柱状图表示独立生物重复实验的平均值±SEM(点状)。p值通过多重未配对t检验计算得出。
(G) 细胞因子抗体阵列展示了对照组和SMARCAL1-KO #1 MDA-MB-436细胞中表达的细胞因子。参考和背景信号展示。
(H) 在(G)实验中,对照组和SMARCAL1-KO #1 MDA-MB-436细胞的细胞因子水平相对于参考信号进行了标准化。
(I) 免疫印迹图显示了对照组和SMARCAL1-KO #1 MDA-MB-436细胞中SMARCAL1、磷酸化STAT1(pSTAT1 Y701)、整体STAT1和tubulin的水平。
(J) RNA测序分析的火山图,比较SMARCAL1-KO #1和对照MDA-MB-436细胞(假发现率[FDR] < 0.05)。红点表示在(D)中的标志基因集中SMARCAL1-KO #1相对于对照细胞表达显著增加的基因。CD274(PD-L1)基因以蓝色标示。
(K) 在经指定siRNA处理的MDA-MB-436细胞、MDA-MB-436对照细胞和SMARCAL1-KO克隆中,PD-L1 mRNA水平的RT-qPCR分析。表达数据的分析和表示方法如(F)所述。p值通过单因素方差分析(one-way ANOVA)计算。
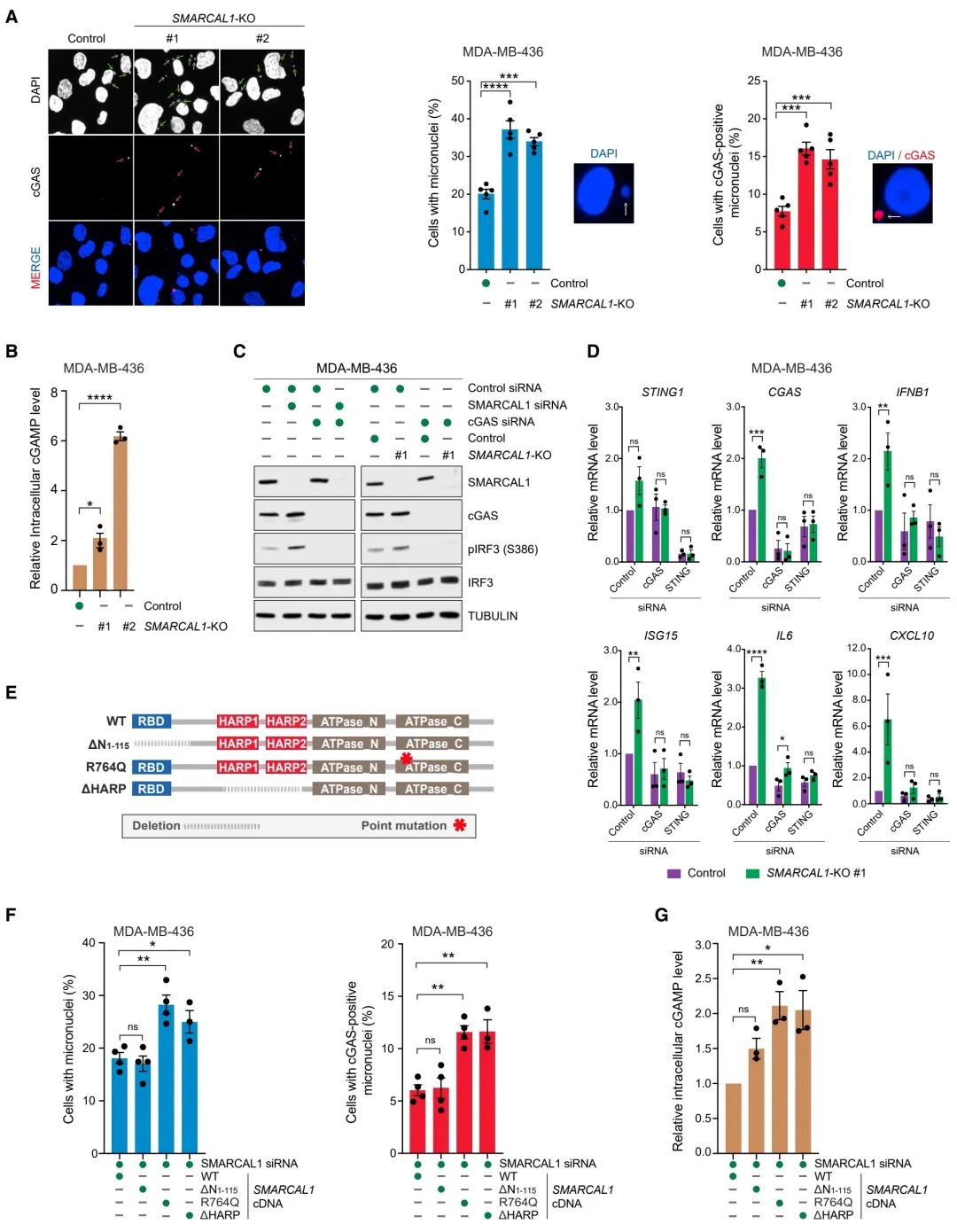
图3:SMARCAL1缺失细胞中固有免疫信号的分析
(A) MDA-MB-436对照细胞和SMARCAL1-KO克隆的DAPI和cGAS染色图像(左图)。显示具有一个或多个微核的细胞百分比(中图)以及带有cGAS阳性微核的细胞百分比(右图)。柱状图代表独立生物学重复实验的平均值±SEM(点状图)。通过单因素方差分析(one-way ANOVA)确定p值。
(B) MDA-MB-436对照细胞和SMARCAL1-KO克隆内部cGAMP水平的相对值。图形表示和统计分析方法同(A)。
(C) 免疫印迹显示经指定siRNA处理的MDA-MB-436细胞、对照组和SMARCAL1-KO #1细胞中SMARCAL1、cGAS、磷酸化IRF3(pIRF3 S386)、整体IRF3和tubulin的水平。
(D) 实时定量PCR(RT-qPCR)分析对照组和SMARCAL1-KO #1 MDA-MB-436细胞在经指定siRNA处理后,选定的干扰素刺激基因(ISGs)和促炎细胞因子的mRNA水平。数据表示与对照siRNA处理细胞相比基因表达的倍数变化。图形表示方法同(A)。统计分析通过多重未配对t检验进行。
(E) 本研究中使用的SMARCAL1及其突变体示意图。
(F) 在用SMARCAL1 WT或指定突变体重构的SMARCAL1缺失MDA-MB-436细胞中,具有一个或多个微核的细胞百分比(左图)或带有cGAS阳性微核的细胞百分比(右图)。图形表示和统计分析方法同(A)。
(G) 与WT相比,在用指定SMARCAL1突变体转染的SMARCAL1缺失MDA-MB-436细胞中内部cGAMP的水平。图形表示和统计分析方法同(A)。
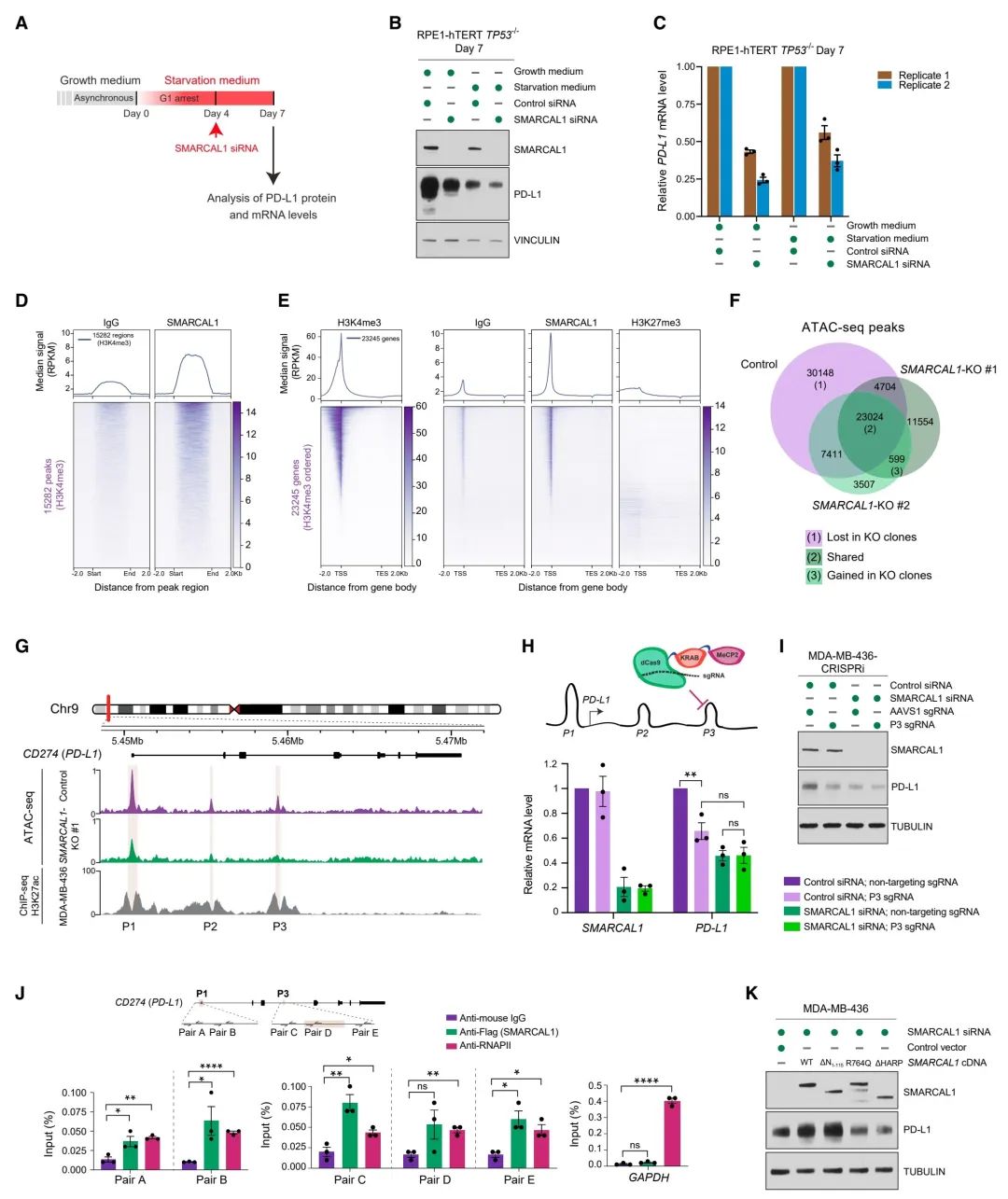
图4:SMARCAL1对PD-L1表达的调节
(A) 用于评估经指定siRNA处理后,复制和非复制RPE1-hTERT TP53-/- 或 U2OS-Fucci细胞中PD-L1水平的实验流程示意图。
(B) 免疫印迹图显示了在培养于生长或饥饿培养基中的RPE1-hTERT TP53-/-细胞中,第4天经指定siRNA处理后,第7天SMARCAL1、PD-L1和vinculin的水平,如(A)所示。
(C) 实时定量PCR(RT-qPCR)分析了如(B)所述培养的RPE1-hTERT TP53-/- 细胞经指定siRNA处理后PD-L1 mRNA水平。数据表示与对照细胞(RPE1-hTERT TP53-/- 细胞经对照siRNA处理后在生长培养基中培养)相比PD-L1表达的倍数变化。柱状图代表技术重复的平均值±SEM(点状图),为两个独立生物重复实验。p值通过单因素方差分析(one-way ANOVA)计算。
(D) 热图显示了MDA-MB-436细胞中通过CUT&RUN技术确定的H3K4me3峰位点的IgG和SMARCAL1信号。RPKM表示每百万映射读数每千碱基的读数。
(E) 热图显示了MDA-MB-436细胞中23,245个编码蛋白的基因在转录起始位点(TSS)前2kb和转录终止位点(TES)后2kb区域内的IgG、SMARCAL1、H3K4me3和H3K27me3 CUT&RUN信号(按基因长度标准化并按H3K4me3信号排序)。
(F) 控制组与SMARCAL1-KO #1-2 MDA-MB-436细胞之间共有和独特的ATAC-seq峰值数量。独特峰值和共有峰值分别代表至少在单个数据集的一个生物重复实验中和所有数据集中存在的峰值。
(G) 控制组与SMARCAL1-KO #1 MDA-MB-436细胞中CD274(PD-L1)位点的标准化ATAC-seq轨迹(每百万计数)。每条轨迹代表三个独立生物重复实验的合并。标识的峰值(P1, 5450318–5450905; P2, 5455316–5455499; P3, 5459366–5459591)被高亮显示。MDA-MB-436细胞中PD-L1位点的H3K27ac ChIP-seq轨迹来自公开可用的数据集(GSE85158)。
(H) 针对P3峰位点使用dCas9-KRAB-MeCP2(顶部)的CRISPRi实验流程示意图。实时定量PCR(RT-qPCR)分析在经指定siRNA处理后并转染了dCas9-KRAB-MeCP2和指定sgRNAs的MDA-MB-436细胞中PD-L1和SMARCAL1 mRNA水平(底部)。数据表示在指定条件下,相对于经对照siRNA处理并转染了dCas9-KRAB-MeCP2和非靶向sgRNA的MDA-MB-436细胞中基因表达的倍数变化。柱状图代表独立生物重复实验的平均值±SEM(点状图)。p值通过多重未配对t检验计算。
(I) 免疫印迹图显示了表达dCas9-KRAB-MeCP2的MDA-MB-436细胞(MDA-MB-436-CRISPRi)在经指定sgRNAs和siRNAs处理后,SMARCAL1、PD-L1和tubulin的水平。
(J) ChIP-qPCR分析SMARCAL1-FLAG和RNA聚合酶II(RNAPII)在P1和P3峰位点的基因组区域或GAPDH启动子作为对照的占据情况。数据代表三次独立实验的结果,并显示技术重复的平均值±SEM。p值通过多重未配对t检验计算。
(K) 免疫印迹显示经SMARCAL1 siRNA处理并补充了野生型或突变SMARCAL1 cDNAs或载体对照的MDA-MB-436细胞中SMARCAL1、PD-L1和tubulin的水平。
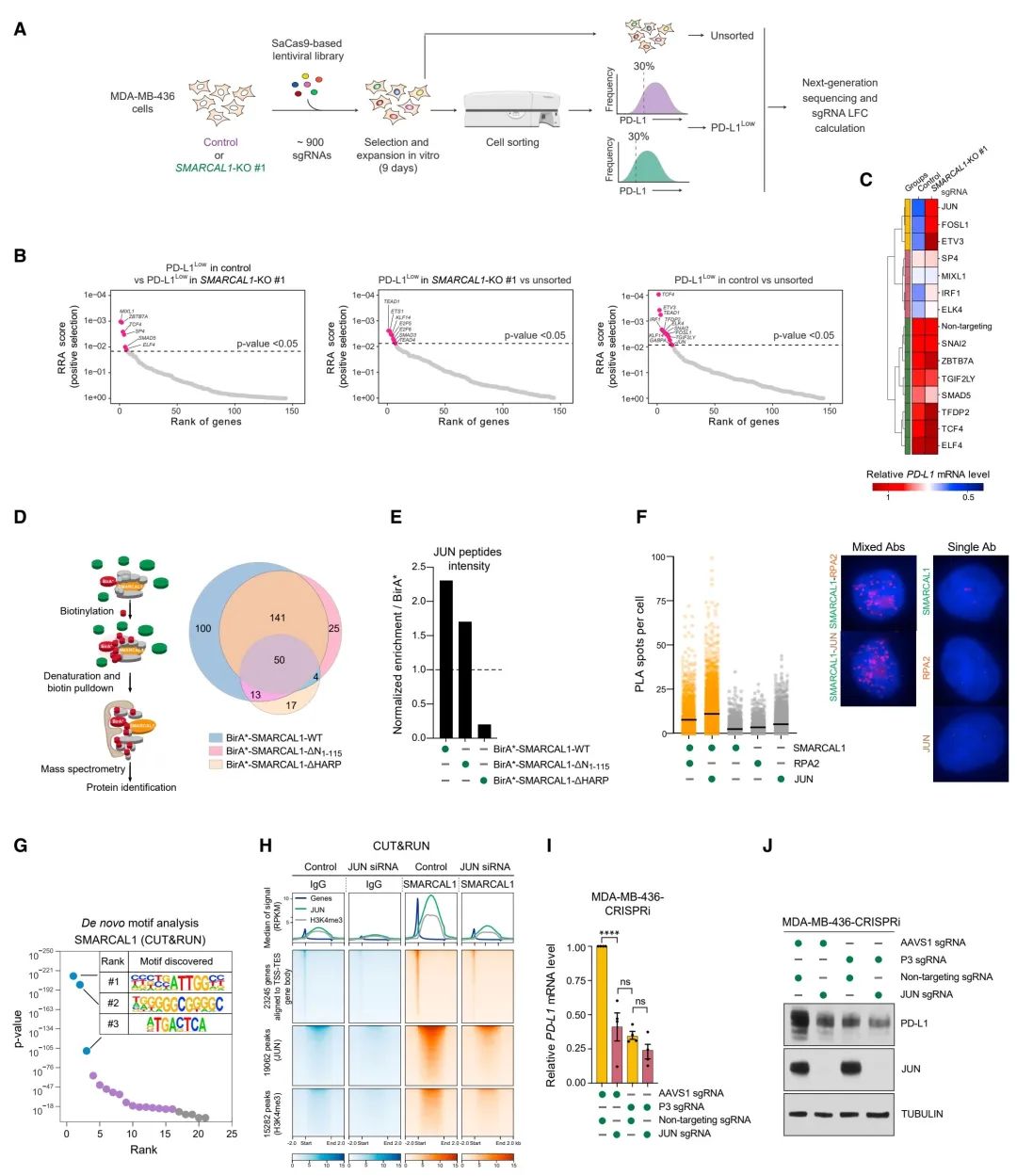
图5:识别与SMARCAL1协同促进PD-L1表达的转录因子
(A) 利用CRISPR筛选技术识别调节SMARCAL1依赖的PD-L1表达的转录因子(TFs)。在选择了转导了sgRNA文库2的对照或SMARCAL1-KO #1 MDA-MB-436细胞后,收集了作为一个混合池(未分类)的细胞或按所示分选的细胞。通过二代测序确定不同细胞群体中sgRNA的丰度。
(B) 根据MAGeCK RRA输出的正向选择分数,对sgRNA文库2靶向的基因进行了排名聚合分数分布(RRA)的显示。粉红点代表与富集的sgRNA相关的基因(正的对数倍数变化和p < 0.05)。
(C) 热图显示了通过RT-qPCR测量的在对照和SMARCAL1-KO #1 MDA-MB-436细胞中转导了指定sgRNA后的PD-L1 mRNA水平的三个生物学重复的平均值。使用欧几里得距离和Ward方法进行聚类分析。详见附表S3。
(D) 用BioID实验识别SMARCAL1相互作用蛋白的示意图。将表达BirA*蛋白或与WT或突变SMARCAL1蛋白融合的BirA*的MDA-MB-436细胞中的生物素化蛋白通过亲和素拉下实验捕获,然后进行质谱分析(左图)。显示了与BirA*对照相比,在指定拉下实验中富集的通过质谱分析鉴定的蛋白质数量(右图)。详见附表S4。
(E) 根据质谱在MDA-MB-436细胞中通过亲和素拉下实验捕获的生物素化蛋白的指定BirA*融合蛋白,得到的JUN肽段强度的标准化富集。数据以相对于BirA*对照的指定拉下实验中JUN肽段强度的变化倍数呈现。
(F) 使用抗SMARCAL1、抗RPA和抗JUN抗体,单独或结合使用,在MDA-MB-436细胞中得到的PLA斑点数量。数据来源于三个独立的生物学重复实验,每个点代表每个细胞的PLA斑点数量。中值已标出。
(G) 使用Homer算法,以SMARCAL1结合的染色质区域为输入峰值,进行de novo发现的基序分析所鉴定的所有基序的p值分布。显示了前三名基序(#1类NFY;#2富含G的基序;#3 AP-1/JUN类)。
(H) 热图展示了MDA-MB-436细胞中,不同JUN耗竭状态下的IgG与SMARCAL1 CUT&RUN信号,这些信号定位在蛋白编码基因的转录起始位点(TSS)前2kb及转录终止位点(TES)后2kb范围内(顶部热图面板)。同样显示了JUN耗竭或未耗竭的MDA-MB-436细胞中,IgG与SMARCAL1信号相对于H3K4me3和JUN峰的定位(底部两个面板)。
(I) 实时定量PCR(RT-qPCR)分析在表达dCas9-KRAB-MeCP2(MDA-MB-436-CRISPRi)和指定sgRNAs的MDA-MB-436细胞中PD-L1 mRNA水平。数据表示指定条件下基因表达的变化倍数,相对于表达非靶向和AAVS1 sgRNAs的MDA-MB-436细胞。柱状图代表独立生物学重复实验的平均值±SEM(点状图)。p值通过单因素方差分析(one-way ANOVA)计算。
(J) 免疫印迹图显示了表达指定sgRNAs的MDA-MB-436-CRISPRi细胞中JUN、PD-L1和tubulin的水平。

图6:针对SMARCAL1缺失的癌细胞的抗肿瘤免疫反应分析
(A) C57BL/6小鼠皮下接种含重组Smarcal1 cDNA或空载体(EV)的B16/F10 Smarcal1-KO细胞后的肿瘤体积分析。图表代表三个独立实验的结果,并显示每组小鼠的平均值±SEM。p值通过未配对t检验计算。
(B) 上述C57BL/6小鼠的生存分析。p值通过对数秩检验计算。
(C) C57BL/6小鼠皮下接种含重组Smarcal1 cDNA的B16/F10 Smarcal1-KO细胞,并伴有或不伴有Cgas基因中断的肿瘤体积分析。图表代表两个独立实验的结果,并显示每组小鼠的平均值±SEM。p值的计算方法同(A)。
(D) 上述C57BL/6小鼠的生存分析。实验和分析方法同(B)。
(E) C57BL/6小鼠接种上述(A)中描述的B16/F10 Smarcal1-KO细胞后肿瘤内淋巴细胞的数量。每个点代表单个肿瘤中每克的浸润淋巴细胞数量。p值通过未配对t检验计算。
(F) C57BL/6小鼠在接种上述(A)中描述的B16/F10 Smarcal1-KO细胞前,经过同种型对照或抗小鼠CD8a抗体处理后的肿瘤体积分析。图形表示和统计分析方法同(A)。详见图S7F和S7G。
(G) 上述(F)中描述的C57BL/6小鼠的生存分析。实验和统计分析方法同(B)。
(H) (NLS)-dsRed-MDA-MB-436对照和SMARCAL1-KO #1细胞,转导PD-L1 cDNA(pHAGE_PD-L1)或空载体(pHAGE_EV),并与或不与人CD8+ T细胞共孵育48小时(T2)的代表性图像。详见图S8D。
(I) 在与活化的CD8+ T细胞共孵育后T2时刻,上述(H)中描述的肿瘤细胞的生长量化。条形图代表在CD8+ T细胞存在时,转导了指定质粒的(NLS)-dsRed-MDA-MB-436对照细胞和SMARCAL1-KO #1细胞相对于没有CD8+ T细胞时的增殖率。柱状图代表三次重复实验的平均值±SD。p值通过两因素方差分析(two-way ANOVA)计算。
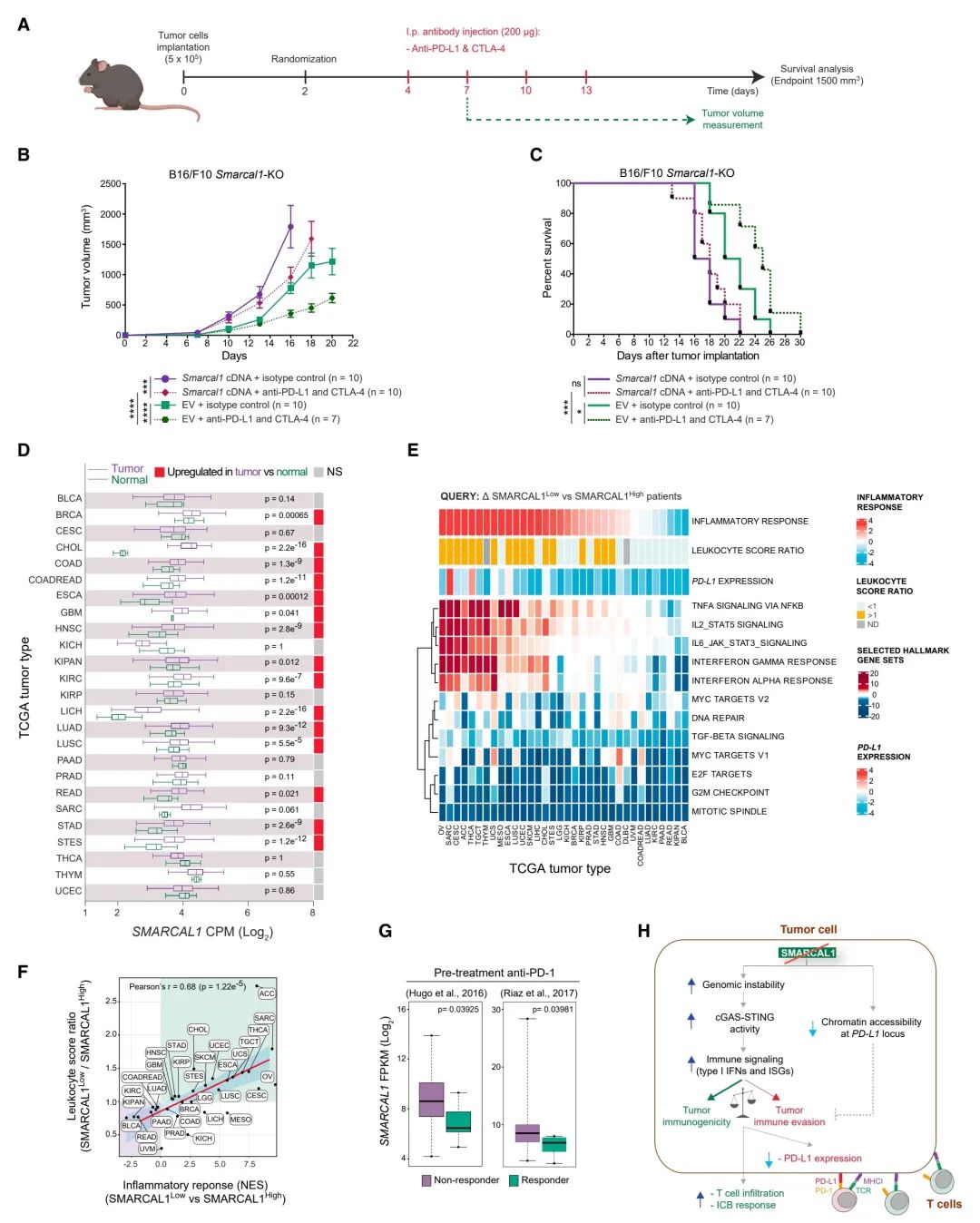
图7:在携带Smarcal1缺失肿瘤的小鼠中进行免疫检查点阻断治疗的效果,以及低SMARCAL1表达癌症患者的临床结果分析
(A) 在注射了重组Smarcal1 cDNA或空载体(EV)的B16/F10 Smarcal1-KO细胞的小鼠中进行免疫检查点阻断(ICB)治疗的示意图。
(B) 在C57BL/6小鼠皮下注射如(A)所述重组的B16/F10 Smarcal1-KO细胞,并随后用同型对照或抗小鼠CTLA-4加抗小鼠PD-L1抗体进行治疗的肿瘤体积分析。数据代表每组小鼠个体的平均值±SEM。p值通过多个未配对t检验计算。
(C) 对(B)中描述的C57BL/6小鼠进行的生存分析。p值通过log rank检验计算。
(D) 在TCGA患者的肿瘤及其相应正常组织中分析指定癌症类型的SMARCAL1表达。p值通过不等方差t检验计算。CPM代表每百万计数。
(E) 在指定的标志基因集(MSigDB)、PD-L1表达和白细胞得分比率中,针对SMARCAL1低表达与SMARCAL1高表达癌症患者的肿瘤背景特异性差异分析。
(F) 对于指定的TCGA肿瘤类型,炎症反应通路的标准化富集得分(NES)与白细胞得分比率之间的皮尔逊相关性分析。
(G) 在接受抗PD-1检查点抑制剂治疗前,非应答组和应答组患者的SMARCAL1水平,来源于参考数据集。p值通过不等方差t检验计算。FPKM代表每百万映射读数每千碱基转录片段的片段数。
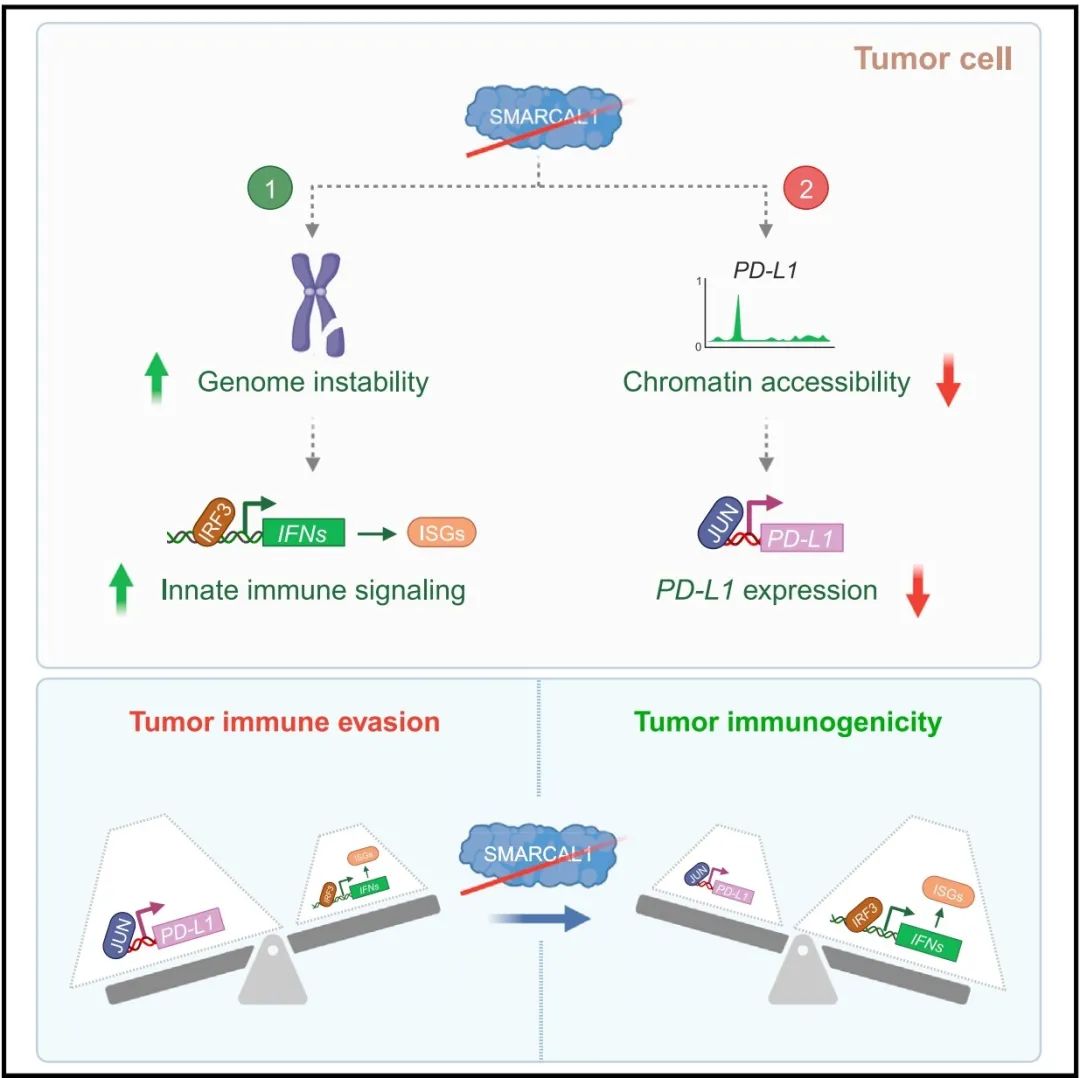
(H) SMARCAL1在调节肿瘤对宿主免疫反应及PD-L1介导的免疫检查点反应中的双重作用模型。
OncoLab公众号已增加期刊查询功能,SCI期刊可查询期刊名称缩写、ISSN刊号、近五年影响因子、最新WOS的JCR分区、最新中科院分区以及中科院期刊预警信息;
中文期刊可检索CSCD核心、北大中文核心、科技核心收录情况;
在公众号对话框发送杂志名称即可快速检索,支持模糊检索,欢迎使用!

关注本号~

加入读者交流群~
(添加请备注单位姓名)

加入知识星球~
点亮赞与在看
让更多人看到
本篇文章来源于微信公众号: OncoLab
 微信扫一扫打赏
微信扫一扫打赏
 支付宝扫一扫打赏
支付宝扫一扫打赏